大家好!打ㄍㄟ厚!胎軋後!各位如果常常追蹤寒波的粉絲團「盲眼的尼安德塔石器匠」,想必對藉由分析古代生物的 DNA,來探討生物演化或是人類遷徙有些認識。但是有人想過這樣的研究是怎麼辦到的嗎?
古代 DNA(ancient DNA, aDNA)研究或古遺傳學(paleogenetics),算是生物學,特別是演化生物學中的研究領域之一。一般指的是最初並非以萃取 DNA 為目標的標本中獲取,再利用特殊方法重建的遺傳資料。藉此重建保存條件不佳的 DNA,並將結果與當代生物的遺傳資訊合併分析,例如,〈冰河期前後智人大移民,其中一群人卻消失了?!〉、〈幾萬年前就是好朋友?在北極發現狗的可能始祖〉都是這類研究的成果。
值得注意的是,雖然我們使用古基因組這樣的詞彙,但這個領域的研究對象並不限於古代的生物,廣義來說,只要樣本的來源不是為了遺傳研究而採集(像博物館中的動植物標本),就算研究的對象不是古代生物,得到的 DNA 依舊可以稱之為古代 DNA。因為 Ancient DNA 資料能具體的呈現研究的生物在過去特定時間點遺傳組成,所以可以讓我們更加了解生物到底是如何變成我們目前看到的模樣。

古 DNA 研究怎麼開始的?
古代DNA研究的起步相當晚,第一個成功的例子在 1984 年,科學家藉由博物館中的動物肌肉標本,重現當時已絕種將近 100 年的斑驢 DNA(quagga,一種斑馬的近親)。隔年,舉世聞名的 Svante Pääbo(沒錯!!就是《尼安德塔人:尋找失落的基因組》的作者帕波)發表了埃及木乃伊的 DNA 片段,立即吸引了大眾的矚目。
對研究者來說,這個全新的資訊來源可以讓他們回答之前無法探討的問題(科學家顯示摩拳擦掌中!!),而流行文化也立即跟上這股風潮,開始想像複製出古生物的可能性,最有名的就是恐龍災難大片《侏儸紀公園》啦!這部經典電影,因為有古生物學家指導此片的拍攝,對大眾傳達了相對正確的演化理論,但是在古代 DNA 研究的概念上(從蚊子中提煉出了恐龍 DNA 那段),卻也低估了古基因組研究過程中的困難。
在早期古基因組重建領域中,帕波是首屈一指的人物。從前述所提到的埃及木乃伊研究開始,到後來讓他揚名立萬的尼安德塔人基因組重建,他的團隊開發了一系列的方法來建立古代 DNA 研究的實驗流程,再配合當時新的 PCR 技術,可以快速擴增目標的 DNA 片段,讓標本的 DNA 重建似乎變得更容易了(詳情請參閱〈尼安德塔人:尋找失落的基因組……科學界30年第一手內幕揭秘〉)。越來越多科學家也加入重建古代DNA的行列,每個人都在比賽誰可以定出最古老的DNA序列……直到那黑暗時期的到來。

一個火雞三明治引發的疑案
這段「黑暗時期」,指的是許多已發表的古代 DNA 研究,最後被證明結果有問題。這包括 1600 萬年前的木蘭化石、琥珀中兩千五百萬到一億兩千萬年前的細菌序列(這情境有沒有一種似曾相識的感覺),都發生實驗結果無法複製的問題。
另一個相當有名的例子發生在 1994 年,有個美國團隊宣稱他們從恐龍的化石,經過 2880 次的 PCR 後,重建出大約 170 個核苷酸長的「恐龍 DNA」。而且這段序列與現存生物的 DNA 相互比較後,發現親緣關係最近的是鳥類,更精確來說是——火雞。這項發現相當符合恐龍與鳥類有最近共同祖先的認知,所以被認為是重要的證據。但後來卻發現這項結果無法被其他研究團隊複製。所謂的「可以複製」,代表他人能夠用作者提供的研究方法和材料重現實驗結果。後續討論中,有人提到「……直到現在,發現恐龍 DNA 的團隊都還在懷疑當時的實驗結果,是否來自他們曾經在實驗室吃過的火雞三明治……」,這些可疑的結果為當時正起步的古代DNA研究蒙上陰影。
各種可能改變DNA的情況
事後科學家從「火雞三明治事件」,與其他失敗的實驗中取得許多教訓。
外來 DNA 汙染
第一個就是實驗汙染。實驗汙染這個問題要跟 PCR 在古代 DNA 上的使用,以及實驗方法一起討論。從生物死亡的瞬間開始,DNA 開始逐漸降解,碎成小段,太短的片段是無法進行 PCR 的,所以樣本中真的可以用來 PCR 的 DNA 片段濃度很低,跟附近環境中的 DNA 數量沒得比。
為了能夠與現在的物種相比較,研究者大多是選用與現生生物類似的基因片段重建。But,人生就是這個 but! 現代生物也有一樣 DNA 啊!畢竟 PCR 酵素不會專挑古代 DNA 來擴增,實驗過程中一開始只要有輕微的環境汙染,現代與與古代樣本間 DNA 數量上的差異就會讓 PCR 的結果完全改變。以「火雞三明治事件」為例,經過 2880 次的 PCR,你想那些輕微的污染會被放大到甚麼程度?

PCR 的技術限制
以 PCR 來重建標本的 DNA 序列還有另一個限制,就是 PCR 一次只能重建一個到數個片段,古代 DNA 往往樣本稀少,如果以建立某個標本完整的一段基因為目標,很可能 PCR 還沒做幾次,材料就用完了。以一段約 600 鹼基對長的基因為例,如果用傳統 PCR 的做法,必須將這段基因分成五個片段 PCR,每次 PCR 的結果都要分別定序,再重新排列組成基因。相當耗費時間與人力。
DNA 的劣化
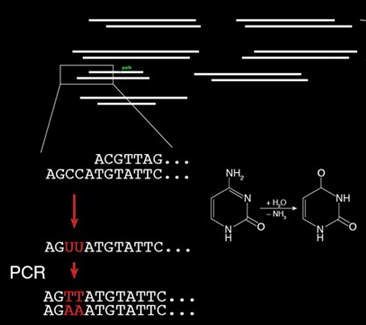
與新鮮的樣本不同,古代 DNA 因為本身的特質跟不良的保存條件,所以萃取的 DNA 往往質量不佳。生物死後,會讓 DNA 的特性跟結構改變,這個機制叫 DNA 死後降解(post-mortem DNA degradation)。除了會發生前述提到的 DNA 分子鍵結斷裂、嘌呤從長鏈分子上脫落、核苷酸的碳環斷裂(我都解釋成DNA逐漸碎掉)之外,也會發生讓不同位置的DNA黏在一起、結構變形阻止 PCR 酵素繼續複製等等千奇百怪的症頭。最有名的例子是 DNA 分子中的胞嘧啶(cytosine)會被轉置成尿嘧啶(uracil)(RNA 的組成分子之一),導致後續 PCR 擴增時原本互補序列上的分子也跟著一起被改變(DNA 錯配,misincoporation),造成定序結果錯誤,這比實驗做不出來更可怕。
但是這些DNA死後降解也能在研究上帶來貢獻,像是其中一種 DNA 損傷的型態——核苷酸鹼基去氨化(DNA deamination),可以當作辨識古代 DNA 的特徵,或是藉由 DNA 損傷在序列中出現的比例,推估到底有多少觀察到的突變是真的因為演化而產生。
影響古代 DNA 保存的因素
古代 DNA 的保存有許多先天限制。雖然直覺是樣本保存的時間越長,DNA的劣化越嚴重,但實際分析發現並不全然如此,在 DNA 開始降解的初期,隨著時間越長,DNA 劣化的程度確實隨之增加。但是當時間對 DNA 降解的影響達到高峰之後,其他的環境因子反而在後續的分解作用中扮演更重要的角色。
舉例來說,氣溫變化就是很重要的影響因子,氣溫越低、以及變化幅度越小,越有益於 DNA 保存,所以現在大部分的古代 DNA 分析樣本都來自於山區、洞穴或是北方永凍層。
另外,早期博物館標本採集時為了保鮮、防止腐敗等原因,也會添加酒精或福馬林等化學藥品(想想那個一趟採集就要花好幾年的時代~),也會影響標本中 DNA 的品質。所以有時就算是一、兩百年的博物館標本,在古代 DNA 重建的難度上還更甚於保存良好的數千年化石樣本。
而不同的分類群間,DNA 分解的效率也有差別,一般來說植物 DNA 的降解速度比動物來的快,研究者推測這可能是不同生物分解時體內微環境不同所造成,但是這方面的研究還沒有定論。
次世代定序是古代 DNA 研究的救星
值得慶幸的是,次世代定序技術(Next-generation sequencing, NGS)使得定序成本和時間明顯降低,更能有效的運用古代 DNA 帶有的資訊。NGS 的其中一個特色是可以廣泛的對全基因組定序,取得大量資料。從〈從3億到1000:3天定序是怎麼辦到的?〉可以知道,現在的分析技術跟過去完全沒辦法比。以前能定序出數個基因片段就偷笑了,現在在 NGS 的加持之下,以一般大腸桿菌基因組為例,跑兩個鐘頭的反應,就可以讀出 2 千 5 百萬個鹼基對。
與 NGS 發展交互影響的是現代資料處理能力的快速進步。電腦科技的進步與普及帶來了大幅增加的資料處理能力,也是古遺傳學得以持續發展的動力。對古代 DNA 的研究者來說,實驗室工作唯一能加強的地方只有對實驗汙染的管理,但是 DNA 降解的問題跟重組都不是在實驗室中進行,而是仰賴計算機的威力,所以很多時候資料處理跟程式運算在研究中反而佔有更高的比重。
古基因組的重組類似拼大張的拼圖,首先你會有一個已知的圖案(reference genome),以重建古基因組來說通常是來自於現生的相似物種,再將 NGS 定序出來的小片段放在合宜的位置。可是現生的物種跟真的要組合的生物還是有一定的差異,就像這拼圖雖然提供給你作品的示範圖片,但是因為內容改版,又跟盒中真的拼出來的結果有微妙的不同。這時就需要借助演算法來推測每個片段該放在拼圖的哪個位置(生物學家向電腦工程師們致上最高的敬意,敬禮!!)。

此外,由於古代 DNA 的品質欠佳,在資料處理過程中需要仰賴程式過濾實驗中汙染、DNA 降解、DNA 錯配所產生的雜訊。資料處理能力的提升也代表突變誤差也可透過數理模型校正。在研究者的努力與新技術的大力加持下,讓數萬年前的尼安德塔人基因組都有機會重見天日,這個領域依舊在蓬勃的發展中。
結論
所以,侏儸紀公園真的會出現嗎?
雖然說這領域的發展日新月異,但樣本的保存期限依舊有物理極限在,而且 DNA 死後降解的速度大多是比理想中來得高的。如同我之前所提到的,過短的 DNA 片段根本無法重組。再加上也不是只要基因組重建完成就可以完整打造出胚胎。我想在可見的將來,我們暫時是看不到恐龍出現在我們眼前啦!!

- 致謝:感謝 Dr. Logan Kistler、神秘友人寒波(盲眼的尼安德塔石器匠格主)與老友林佩蓉對專業內容與文章結構上的建議與修改。本文為作者參與歐盟居禮夫人人才培育計畫創新訓練網絡(Innovative Training Networks, Marie Curie Actions)之子計畫 MicroWine 所撰寫。
參考資料:
- Rizzi, Ermanno, et al. “Ancient DNA studies: new perspectives on old samples.” Genet Sel Evol 44.1 (2012): 21.
- Poinar, Hendrik N., and A. Cooper. “Ancient DNA: do it right or not at all.” Science 5482 (2000): 1139.
- Hoekstra, Hopi E., and Catherine L. Peichel. “Genes, Genomes, Phenotypes.” The Princeton Guide to Evolution (2013): 363.