- 解讀丨陳柱成、嚴麗娟(清華大學)
- 責編丨迦漵
核小體是真核生物染色質的基本單位,其核心由~147 bp DNA纏繞組蛋白八聚體形成。染色質重塑複合物(Chromatin remodeling complexes,CRC) 利用 ATP 的能量移動核小體在基因組上的位置和組成成分,在控制染色質結構、調節基因轉錄等方面具有重大作用。根據結構和功能的特點,染色質重塑複合物可以分四大類:SWI/SNF、CHD、ISWI和INO80。1
這些分子機器的運行機理,即如何利用 ATP 水解的能量推動核小體移動和組蛋白交換,一直是一個未解的科學問題。利用冷凍電鏡技術,近年來對於這個問題的解答有了突飛猛進2-5。
2018年10月12日,來自英國帝國理工學院的 Dale B. Wigley 課題組和 David S. Rueda 課題組合作在 Science 雜誌發表長文,解析了酵母 SWR1 染色質重塑複合物結合核小體的三維結構,並結合單分子FRET實驗,揭示了 SWR1 催化組蛋白變體替換的動力學過程6。
SWR1屬於INO80染色質重塑家族,其主要功能是催化 H2A-H2B 與 H2A.Z-H2B 的替換。H2A.Z是H2A的變體,富集在啟動子區域,也能抑制異染色質向常染色質的延伸7。在染色質的特定位置或特定時期進行組蛋白變體的替換可以改變染色質結構或招募染色質調控因數,是維持不同染色質狀態所必需。與其它染色質重塑蛋白不同,SWR1不能移動核小體。因此,SWR1 如何利用 ATP 水解的能量催化 H2A-H2B 交換尤其讓人困惑。
為了研究 SWR1,Wigley 課題組沒有採用常規的提取內源蛋白的方案,而是利用昆蟲細胞,體外表達重構了十四個亞基的完整酵母 SWR1複合物。通過相同的方法,Wigley課題組今年初解析了INO80複合物的結構4。
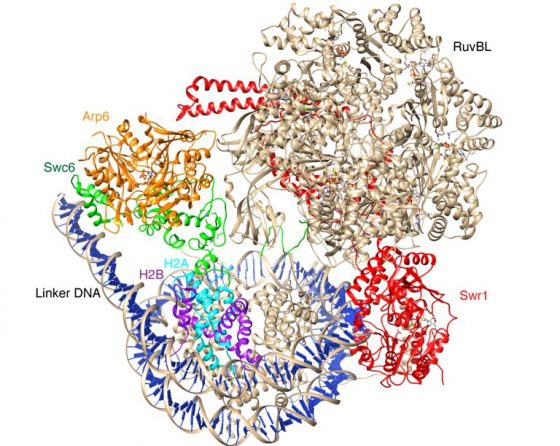
SWR1複合物全酶的核心催化亞基是Swr1,它的ATPase活性是組蛋白替換的關鍵。Swr1結合核小體的SHL2位置,這與Ino80 不同,但與其它的染色質重塑蛋白類似。更重要的是,Swr1的結合使DNA在SHL2處產生1 bp凸起(bugle)。為了形成這個凸起,1 bp DNA從進口端被抽進核小體內部。 由於1bp DNA的位移,Swr1的N端結構域和核小體的另一圈DNA之間相互作用加強,限制了核小體的進一步滑動,這是SWR1不具備催化核小體滑動的可能機制。需要特別指出的是,Swr1處於ADP-BeF3 (ATP類似物) 狀態。已知的其它染色質重塑蛋白沒有發現在ATP結合狀態下引起1 bp DNA位移3-5。這種異常的局部DNA移動也可能是SWR1沒有真正的核小體滑動活性的原因。
複合物全酶中另一個重要的核小體結合元件是Arp6-Swc6異源二聚體。它們通過全酶中的RuvBL亞基(六聚體)與Swr1 作用,穩定了整個複合物的構象。Arp6-Swc6結合進口端DNA,使得該DNA偏離了常規位置~65°,暴露大概2.5圈核小體DNA。突變和生化實驗(體外表達重構體系的一大優勢)表明Arp6-Swc6 是SWR1組蛋白替換活性的關鍵。與INO80一樣,因為失去了與進口端DNA的作用,H2A-H2B也相應地暴露。這也許是SWR1與INO80共同具有組蛋白替換活性的機理。
作者進一步採用單分子FRET手段監測了SWR1解開核小體末端DNA纏繞的過程。實驗表明在加入SWR1複合物和ATP後,末端DNA顯著偏離常規位置;而加入ATPγS 和ADP•BeF3也捕捉到DNA解開的現象,這說明SWR1誘導的核小體DNA去纏繞依賴ATP的結合而不是水解。
儘管SWR1在ADP狀態下與核小體的作用方式目前仍不清楚,Wigley這個工作的發表使我們理解SWR1催化組蛋白替換的機制又前進了一大步。Dale Wigley是研究DNA解旋酶、滑移酶的大師,早在 1999 年通過研究PcrA, 提出了 DNA滑移酶的教科書級的inchworm(尺蠖,下圖)運動模型8。這個模型為理解染色質重塑蛋白的工作機理提供了基礎。

相對於INO80 和 SWR1 複合物,人們對 SWI/SNF 的研究,尤其是哺乳類動物SWI/SNF (mSWI/SNF),有點滯後。mSWI/SNF 是細胞生長、發育和分化等過程所必需的生物大分子,約 20%的人類癌症中發現mSWI/SNF 的突變9。
近日,美國哈佛大學Cigall Kadoch課題組在Cell上發表了題為Modular Organization and Assembly of SWI/SNF Family Chromatin Remodeling Complexes的研究長文,揭示了mSWI/SNF複合物的模組組織和組裝通路,並分析了人類疾病相關的突變影響mSWI/SNF的分子機制。
人源 SWI/SNF 複合物由超過 20 個基因編碼,基於特定的人體組織和發育時期,複合物採用不同的蛋白亞基(paralogs和variants)和組裝形式,其結構組成紛繁複雜,難於一一辨認。早在 2013 年,Kadoch還是博士生的時候,她通過生化和質譜手段,鑒定了不同細胞系的mSWI/SNF組成10。
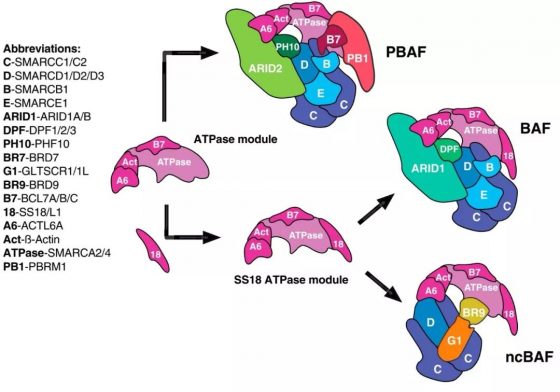
在當前的研究中,Kadoch課題組進行了更全面系統的工作,優化了一套穩定的mSWI/SNF複合物純化策略。他們利用CRISPR-Cas9在HEK293T細胞系中mSWI/SNF相關亞基上導入HA標籤,通過一系列蛋白純化手段,得到了野生型和多種突變體mSWI/SNF複合物。通過分析不同的複合物組成和交聯質譜資訊,他們鑒定出HEK293T細胞系中三種mSWI/SNF複合物,分別是BAF(BRG1/BRM-associated factor)、PBAF(polybromo-associated BAF)和 ncBAF(non-canonical BAF,一種新發現的複合物)(下圖)。
為了更細緻地分析各亞基之間的相互作用以及 mSWI/SNF複合物組裝的過程,作者敲除不同的亞基,純化得到不同的複合物,進行交聯質譜和網路模組分析。最後,作者提出了mSWI/SNF的組裝通路:SMARCC1/SMARCC2和SMARCD1是mSWI/SNF複合物的初始平臺(initial core);initial core、SMARCB1和SMARCE1構成的亞複合物定義為BAF複合物的核心模組(BAF core module)。ARID1和BAF core module形成亞複合物(ARID1/BAF core),然後與DPF2結合,加上ATPase module(含SS18)完成BAF組裝;而ARID2與BAF core 形成亞複合物(ARID2/PBAF core),結合BRD7 和PHF10之後招募ATPase module(不含SS18),最後結合 PBRM1完成組裝過程。另一方面,GLTSCAR1/1L BRD9和BAF core 結合, 形成ncBAF core module,合併BRD9和ATPase module(含SS18)形成ncBAF複合物。
這個BAF複合物的構成與較早報道略有不同。其中,BRD9在較早研究中是BAF複合物的一個亞基,現在劃歸到ncBAF中;另外,BCL11似乎在HEK293T細胞系的BAF複合物中沒有被發現,而在T-細胞的BAF複合物中存在【10】。這些差異正好體現了mSWI/SNF研究的挑戰性。
作者結合含突變位點的BAF複合物的細胞和生化分析,根據勾勒出的mSWI/SNF複合物的組織輪廓,解釋了一些高頻錯義或無義突變影響mSWI/SNF複合物的組裝通路和各亞基之間的穩定性進而導致人類癌症及其他相關疾病的可能機理。總之這項研究對於後續 mSWI/SNF複合物的結構和功能研究提供了很有參考價值的框架。

Cigall Kadoch是一個非常出色的科學家,一直致力於研究哺乳動物 SWI/SNF 的功能與人類癌症的關聯。據說,她在哈佛大學尋找博士後工作時,因為實在太優秀了,沒有給她博士後崗位,而是直接讓她當獨立PI。2015 年,Cigall Kadoch當選MIT技術評論Innovator Under 35。
注:清華大學生命科學學院陳柱成課題組主要運用結構生物學的方法從事染色質生物學相關研究,近年來連續在Nature上發表兩篇研究論文闡述染色質重塑的機理11,12。
BioArt補記
就在英國帝國理工學院的 Dale B. Wigley 課題組和 David S. Rueda 課題組合作在 Science 雜誌發表長文,解析了酵母SWR1染色質重塑複合物結合核小體的三維結構之後一周左右,來自復旦大學生物醫學研究院徐彥輝課題組在 Cell Research 雜誌上發表了題為 Cryo-EM structure of human SRCAP complex的研究論文13,首次解析了 4埃解析度的人源 SRCAP 複合物(對應酵母中 INO80 染色質重塑複合物)冷凍電鏡結構,為後續深入研究核小體置換機制奠定了基礎。
SRCAP-C為超大複合物,分子量約為 1 MDa(1000KDa),包含10個組分(SRCAP, DMAP1, YL1, RUVBL1, RUVBL2, ACTL6A, ARP6, ACTIN, GAS41 and ZNHIT1)。結構和生化研究表明在SRCAP-C中SRCAP不僅具有ATP解旋酶酶活性,還作為支架蛋白幫助複合物組裝。其中間部分貫穿RUVBL1-RUVBL2六聚體,其N端和C端分別連接多個SRCAP複合物組成蛋白。 RUVBL1-RUVBL2形成的六聚體為穩定核心,從底部兩側延伸出來SRCAP解旋酶結構域及Actin相關蛋白等模組。這兩個相對應的模組具有DNA結合的表面特徵,很可能為核小體結合位點。研究工作根據SRCAP同家族蛋白INO80與核小體複合物的結構,類比了SRCAP與核小體相互作用方式,為後續深入研究核小體置換機制奠定了基礎。
參考文獻
- Clapier, C. R. & Cairns, B. R. The biology of chromatin remodeling complexes. Annu. Rev. Biochem. 78, 273-304 (2009).
- Liu, X., Li, M., Xia, X., Li, X. & Chen, Z. Mechanism of chromatin remodelling revealed by the Snf2-nucleosome structure. Nature 544, 440-445 (2017).
- Farnung, L., Vos, S. M., Wigge, C. & Cramer, P. Nucleosome-Chd1 structure and implications for chromatin remodelling. Nature 550, 539-542 (2017).
- Ayala, R. et al. Structure and regulation of the human INO80-nucleosome complex. Nature 556, 391-395 (2018).
- Eustermann, S. et al. Structural basis for ATP-dependent chromatin remodelling by the INO80 complex. Nature 556, 386-390 (2018).
- Willhoft, O. et al. Structure and dynamics of the yeast SWR1-nucleosome complex. Science 362(2018).
- Talbert, P. B. & Henikoff, S. Histone variants on the move: substrates for chromatin dynamics. Nat. Rev. Mol. Cell Biol. 18, 115-126 (2017).
- Velankar, S. S., Soultanas, P., Dillingham, M. S., Subramanya, H. S. & Wigley, D. B. Crystal structures of complexes of PcrA DNA helicase with a DNA substrate indicate an inchworm mechanism. Cell 97, 75-84 (1999).
- Hodges, C., Kirkland, J. G. & Crabtree, G. R. The Many Roles of BAF (mSWI/SNF) and PBAF Complexes in Cancer. Cold Spring Harb Perspect. Med. 6(2016).
- Kadoch, C. et al. Proteomic and bioinformatic analysis of mammalian SWI/SNF complexes identifies extensive roles in human malignancy. Nat. Genet. 45, 592-601 (2013).
- Yan, L., Wang, L., Tian, Y., Xia, X., & Chen, Z. (2016). Structure and regulation of the chromatin remodeller ISWI. Nature, 540(7633), 466.
- Liu, X., Li, M., Xia, X., Li, X., & Chen, Z. (2017). Mechanism of chromatin remodelling revealed by the Snf2-nucleosome structure. Nature, 544(7651), 440.
- Feng, Y., Tian, Y., Wu, Z., & Xu, Y. (2018). Cryo-EM structure of human SRCAP complex. Cell research, 1.
【原文属于BioArt】BioArt,一心关注生命科学,只为分享更多有种、有趣、有料的信息。投稿、合作、转载授权事宜请联系微信ID:fullbellies 或邮箱:sinobioart@bioart.com.cn