本文由台大化學蔡蘊明教授譯自諾貝爾化學獎委員會公佈給大眾的新聞稿(2014/10/9)
PanSci 編輯部轉載並編輯修改自台大化學網站。
艾瑞克・貝齊格(Eric Betzig),史蒂芬・海爾(Stefan W. Hell)以及威廉・莫納(William E. Moerner)等三人得到了2014年的諾貝爾化學獎,這是因為他們越過了一個科學上設想的限制,也就是光學顯微鏡的解析度永遠無法比 0.2 微米更精確。但如今,利用分子的螢光,科學家現在可以監看在細胞內部分子之間的相互作用;他們可以觀察與疾病相關的蛋白質之聚集,也可以在奈米的尺度裡追蹤細胞分裂。
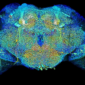
紅血球細胞、細菌、酵母菌細胞以及游動精子:當科學家在十七世紀第一次開始在顯微鏡下研究活體組織時,一個新的世界在他們的眼前打開。這是微生物學出世之際,從此之後,光學顯微鏡成為生命科學家工具箱裡面最重要的工具之一。其它的顯微鏡術,例如電子顯微鏡,其所需的準備方法最終會殺死細胞。
發亮的分子越過了物理的屏障
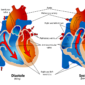
然而,有一段很長的時間,物理條件限制了光學顯微鏡所能解析的結構的大小。在1873年,顯微鏡學家恩斯特・阿貝(Ernst Abbe)發表了一個方程式,證明了光學顯微鏡的解析度是如何受到光的波長,以及一些其它的因素所限制。這導致科學家在二十世紀的大半時間裡,相信光學顯微鏡是永遠無法用來觀察那些比所用的光之波長的一半還小的物體,也就是 0. 2微米 (200奈米;微米 = 10-6 米 = 10 3 奈米) (圖一)。在這樣的狀況下,細胞裡一些胞器的輪廓,例如細胞的發電機粒線體,雖可以看到,但是幾乎不可能分辨更小的物體,因此如果想要追蹤細胞裡蛋白質分子之間的相互作用,就無法做到,這好比能看到一個城市的建築物,但卻無法看出市民如何的生活,和如何為其生存而努力。為了瞭解一個細胞如何的運作,你必須能追蹤個別的分子如何的工作。
儘管阿貝的方程式依然成立,但繞射極限的障礙仍被克服了。艾瑞克・貝齊格,史蒂芬・海爾以及威廉・莫納等三人之所以獲得2014年的諾貝爾化學獎,就是因為他們利用螢光分子,將光學顯微鏡帶進了另一個境界。理論上,不再存在有太小而無法觀察的結構。就結果而言,光學顯微鏡變成了奈米顯微鏡。
如何規避阿貝繞射極限的故事,要分成兩條路線來說;兩個基於不同的原理所各自獨立發展出的方法,都獲得成功。讓我們回溯到1993年,在芬蘭西南部的一個學生公寓裡,史蒂芬・海爾在翻閱一本量子光學的教科書時,得到了一個很棒的點子。
對阿貝繞射極限的青春叛逆面對了懷疑
自從海爾在1990年從德國海德堡大學取得博士學位之後,他就一直在尋找方法,來規避阿貝在超過一個世紀以前所訂下的限制。挑戰一個已經建立的理論,這樣的想法雖很誘人,但是在德國的資深科學家們,以懷疑面對他的熱情,導致了海爾往寒冷的北方尋找庇護所。一位在芬蘭特爾庫(Turku)大學研究螢光顯微鏡術的教授,給了海爾在其研究小組工作的一個職位。海爾相信一定有一個機會能夠克服阿貝的繞射極限,而當他讀到那本量子光學課本裡面「受激放射」的字語時,在他的腦海裡浮現了一個新的想法:「在那個瞬間,曙光在我腦際出現,我終於找到一個實際的觀念來追求一條真正的線索。」這是他於2009年自己的說明 ,讓我們進入他的想法一探究竟。
解答:用奈米大小的手電筒掃描樣品
在特爾庫大學,海爾在進行稱為螢光顯微鏡術的研究,那是一種利用螢光分子來讓細胞顯像的技術。舉例來說,他們可以使用只與特定細胞DNA偶合之專一螢光抗體,再用一個短暫的脈衝光來激發螢光抗體,這可以讓抗體短暫並持續一段時間。而如果抗體的確與DNA偶合,它們就會在細胞當中放瑩光,因為DNA是塞在細胞核裡面的。利用這個方法,科學家們可以看到某些分子的位置,但是他們只能定出一群聚集在一起的分子之位置,例如一些糾纏在一起的多股DNA,但是因為解析度太低,而無法分辨單股的DNA,這就好像你可以看到一卷紗線,但卻無法看出紗線是如何纏繞的。
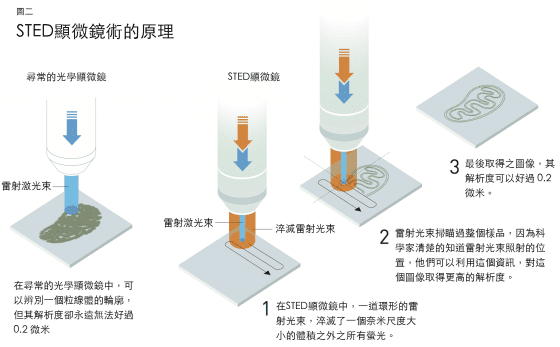
當海爾讀到受激放射時,他體認到應該可以設計一種範圍為奈米大小的手電筒,能夠對著樣品以一次一個奈米的方式掃描。利用受激放射,科學家們可以將分子的螢光淬滅(quench),當他們將一道雷射光束照在那些發光的的分子上時,它們會立刻失去能量而變暗。在1994年,海爾發表了一篇論文概略說明了他的想法,他規劃的方法稱為受激放射消去法(stimulated emission depletion,簡稱STED),利用一道脈衝光將所有的螢光分子激發(開始發光),同時利用另一道脈衝光將所有的螢光分子淬滅,但是只有在中間的一個奈米尺度大小的體積之內除外(圖二),因此只會取得在這個體積之內的螢光。透過對樣品的掃描以及同時對光線強度的測量,就可以取得一張清楚的圖像。每一次被容許放出螢光的體積愈小,最後得到的影像解析度就愈高,因此在理論上,光學顯微鏡在解析度方面就不再有限制了。
在德國發展頭一個奈米手電筒
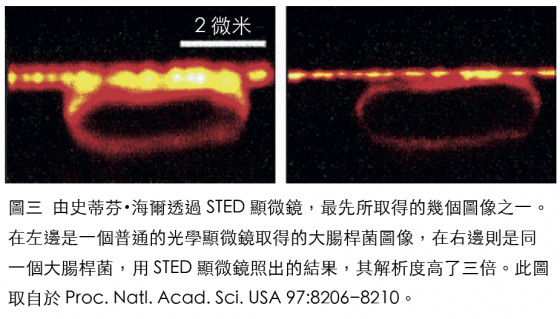
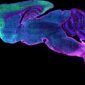
海爾的理論文章並未立刻的激起一場騷動,但是的確有趣到讓海爾在位於哥廷根的馬克斯・卜蘭克生物物理化學研究所,得到一個職位。在接下來的數年裡,他讓自己的想法開花結果;他設計了一個STED顯微鏡,於2000年,已經能夠展示真的可以實際的運用他的想法,其中之一是用來取得一張大腸桿菌的圖像,並具有用光學顯微鏡從來無法達到的解析度(圖三)。
STED 顯微鏡從收集一大堆很小的體積所放出的光,然後集合成一張整體的圖像,相對的比較,另一種原理也得到了成功,那被稱為單分子顯微鏡術,需要將許多張圖像重疊在一起。艾瑞克・貝齊格與威廉・莫納(大家都用W. E.稱呼他)各自獨立的,以不同的基礎觀念切入,促成這項技術的發展。這項技術的基礎,是在莫納成功的觀測到一個小的螢光分子時所奠定。
W. E. 莫納 ― 首先觀測到單一的螢光分子
在大部分的化學方法中,例如量測吸收和螢光,科學家們是同時觀察上百萬的分子,在這些實驗中所得到的結果,反映的只是一種典型平均化的分子表現,但科學家們不得不接受這種困境,因為沒有別的可能性。不過有很長的一段時間,他們夢想著能夠量測每一個單一的分子,因為有愈豐富愈詳盡的資訊,就愈可能去瞭解譬如疾病是如何的發展。
在1989年,莫納成為全球第一位科學家能夠量測單一分子對光的吸收,那是一項具有關鍵性的成就。當時他正在位於美國加州聖荷西的IBM研究中心工作,那個實驗打開了一扇通往新未來的大門,並且啟發了許多化學家將注意力轉移到單分子的身上,其中之一就是艾瑞克・貝齊格,接著會在稍後說明他的成就。
八年之後,莫納朝單分子顯微鏡邁出了第二步,那是運用之前諾貝爾獎在2008年所表彰過的綠色螢光蛋白質(GFP)。
分子大小的燈一開一關
在1997年,莫納進入了在加州大學的聖地牙哥分校,那正是後來獲得諾貝爾桂冠的錢永健所在的學校,當時錢永健正嘗試要讓GFP放出像彩虹般的各種螢光。這個綠色螢光蛋白質是從一種螢光水母身上分離出來的,它的好處在於能讓細胞裡面的其它蛋白質顯像。科學家們先利用基因科技,將綠色螢光蛋白質偶合到其它的蛋白質上,那綠色的螢光就會暴露出這個被標記的蛋白質位在何處。
莫納發現有一種GFP可隨意點亮或關掉,當他用488奈米波長的光去激發蛋白質的時候,蛋白質就開始發出螢光,但一個短暫的時間之後就會熄滅,在這之後無論他再用多強的光去照射這個蛋白質,它也不會發光,不過他後來發現當光的波長改為405奈米時,這個蛋白質就會恢復生機,當蛋白質重新活化後,它又會放出488奈米波長的螢光。
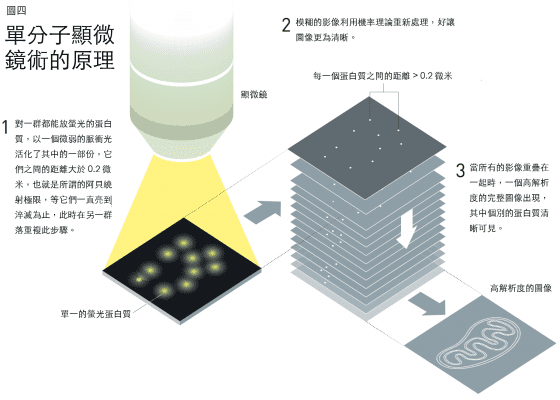
莫納將這些可被激發的蛋白質均勻的散佈在一個膠質內,讓每個蛋白質之間的距離大於0.2微米的阿貝繞射極限,因為它們稀疏的散開來,一個普通的光學顯微鏡就可以區辨每一個發亮的分子 ― 它們就好像一堆具有開關的小燈泡,這項結果發表在1997年的“自然”期刊上。
透過這個發現,莫納展示了可以透過光學的方式,控制單一分子們的螢光,這解決了一個貝齊格在兩年之前所想到的問題。
對學術感到疲乏 ― 但仍爲阿貝的繞射極限而著迷
與海爾一樣,貝齊格也爲了越過阿貝繞射極限的想法而著迷。在1990年代初期,他正在美國紐澤西州的貝爾實驗室,研究一種新的光學顯微鏡術,稱為近場顯微鏡術。在此法中,光線是從一個非常薄的尖端所釋出,這個尖端與樣品之間的距離只有幾個奈米,雖然這種顯微鏡術也可以克服阿貝繞射極限,但是此法具有一些主要的弱點,舉例來說,因為放出的光範圍太短(只能深入約一百奈米),以至於無法看到細胞表面之下的結構。
貝齊格在1995年得到一個結論,那就是近場顯微鏡術無法更進一步的改善,此外他在學術界感覺不太自在,因此決定結束他的研究生涯;即便不知下一步要何去何從,他毅然辭職,但是阿貝繞射極限仍在他的心中。步行在一個寒冷的冬天裡,他想到了一個新的點子;是否可能用具有不同性質的分子,那些發出不同顏色之螢光的分子,來克服阿貝繞射極限?
貝齊格已經能用近場顯微鏡術觀測到單分子的螢光,與許多人一樣,貝齊格受到莫納的啟發,他開始仔細考慮,如果使用幾種會放出不同螢光的分子,例如紅色、黃色和綠色,是否可以利用普通的光學顯微鏡得到相同的解析度。他的點子是讓顯微鏡每一次用不同顏色的光來記錄影像,如果同一種顏色的分子都是均勻的散佈,而且相互之間的距離大於阿貝繞射極限的規範,它們的位置將可精確的決定。接著當這些影像重疊起來時,完整的圖像將具有遠超過阿貝繞射極限的解析度,紅色、黃色和綠色的分子雖然相互的距離只不過幾個奈米,但仍能區別,如此就能克服阿貝繞射極限。不過,仍有一些實際的困難,例如缺乏那些具有不同光學性質之分子,其差異要大到足以相互區別。
在1995年,貝齊格在 Optical Letters 這份期刊上發表了上述想法之理論,隨即離開了學術界,並進入了他父親開的公司。
被綠色螢光蛋白質引誘回到顯微鏡術
貝齊格完全的脫離學術界,已經有許多年了,但是有一天,一個對科學的渴望突然又復甦了。回顧科學文獻時,他第一次看到綠色螢光蛋白質的論文,體認到有一個蛋白質,能讓其它的蛋白質在細胞內顯像,活化了貝齊格對如何克服阿貝繞射極限的想法。
真正的突破發生在2005年,當時他偶然發現到那種可以隨意活化的螢光蛋白質,很類似那些莫納在1997年,於單分子的層次所觀察到的螢光蛋白質。貝齊格知道,這個分子正是可以實現他在十年前所想到的那個主意,所需要的工具。這種螢光分子並不需要具有不同的顏色,它們還是可以在不同的時間發出螢光。
藉著影像的重疊超越阿貝繞射極限
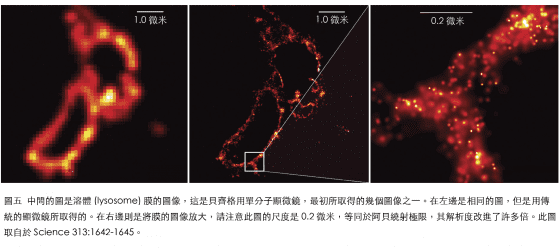
只不過一年之後,與研究可激發螢光蛋白質的科學家合作,貝齊格展示了他的想法的確可以付諸實現。在一些例子當中,他們將會發光的蛋白質接在溶體(lysosome)的膜上面,溶體是細胞裡的回收站,現在用一道脈衝光來激發出蛋白質的螢光,因為使用的脈衝很弱,所以只能讓部分的分子開始發出螢光,由於它們的數目很少,幾乎所有發光分子之間的距離均大於0.2微米的阿貝繞射極限,因此每一個發光的蛋白質之位置都可以在顯微鏡下登錄。一會兒之後,當螢光消失時,他們重新激發另一組蛋白質,同樣的,使用的脈衝弱到只能讓部分的分子發出螢光,同時這一組圖像被登錄下來,這個步驟一直不斷的重複。
當貝齊格將所有的影像重疊起來時,得到了一張溶體膜的超高解析圖像,它的解析度遠遠的超過了阿貝繞射極限。接著,貝齊格將這一份開創性的工作,於2006年發表在“科學”期刊上。
這幾位得獎者仍企圖在描繪生命最深層的奧秘
這些由艾瑞克・貝齊格,史蒂芬・海爾以及威廉・莫納等三人所開發的方法,發展出了幾個現在爲全世界各地所使用的奈米顯微鏡技術。這三位得獎者仍然活躍在這個不僅龐大,而且一直在增長的科學社群中,將創新的矛頭對著奈米顯微鏡術的領域,當他們將功能強大的奈米顯微鏡瞄準在生命中最小的零件時,他們也同時取得了最尖端的知識。史蒂芬・海爾爲了對腦突觸有更好的瞭解,窺探了活的神經細胞內部;威廉・莫納研究了與杭丁頓氏症(舞蹈症)有關的蛋白質;艾瑞克・貝齊格追蹤了在胚胎中細胞的分裂,這些只是眾多例子當中的幾個。有一件事情是肯定的,2014年的諾貝爾化學桂冠得主們,對發展人類最重要的知識,已經奠定了基石。
*特別感謝現於美國德州農工大學攻讀博士的曹一允(我2008年的專題生)熱血相挺,幫我將圖片中文化;另外感謝現於本系李弘文教授實驗室,攻讀碩士學位的林宇軒幫我校稿。多年來幫我將譯文置於台大化學系網頁的黃俊輝先生業已退休,感謝接替他的蔡明軒幫忙。
編按:蔡教授歷年翻譯的諾貝爾化學獎得主貢獻簡介