本文轉載自中央研究院研之有物,泛科學為宣傳推廣執行單位
- 採訪編輯|張容瑱、美術編輯|林洵安
「以前只會吃香蕉,不會特別關心香蕉。」中研院農業生物科技研究中心陳荷明副研究員,過去研究主題無關香蕉,也沒有想過自己有一天會研究香蕉,但有鑑於香蕉黃葉病影響甚鉅,加入了研究的行列。她帶領團隊運用基因序列資料分析,找到鑑定台灣抗黃葉病香蕉品種的分子標誌,保障台灣香蕉的品種專利權。跟著研之有物一起來了解!
抗黃葉病品種的分子標誌
處處買得到的香蕉,好吃、便宜又很方便,剝開皮就可以吃,兩三口就吞下肚,因為果肉裡沒有籽,不需要一邊吃一邊吐籽…… 等等!你有沒有想過:香蕉沒有籽,怎麼繁衍下一代?

圖片來源│iStock
香蕉,其實有籽……
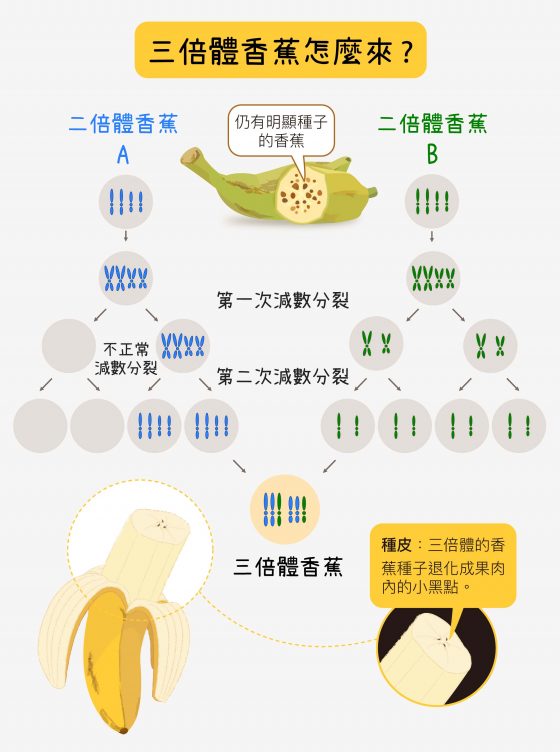
切開香蕉,果肉中軸附近有一些黑點,有些比較明顯,有些幾不可見。這些黑點正是香蕉已退化或發育不良的種子。因為市面上的食用香蕉大多是三倍體,也就是有三套染色體的華蕉。三倍體的香蕉會開花、會結出果實,但沒辦法發育出正常的種子。
人類是二倍體,有兩套染色體,一套來自父親、一套來自母親。像香蕉這樣染色體超過兩套的,稱為「多倍體」,這在動物界很少見,在植物界卻相當普遍,像小麥是六倍體,草莓則是八倍體。由於多倍體結的果實通常比較大,而且相較於二倍體來說,有更多染色體可儲存基因,具備比較多樣的性狀,在農業上有許多應用價值。而且三倍體的香蕉種子會退化,能讓果肉吃起來柔軟滑順。
沒有種子,怎麼繁殖?香蕉採用「營養繁殖」,利用從塊莖冒出來的「吸芽」產生新的植株。栽種時,農民可直接挖取母株旁邊由吸芽發育長成的小香蕉樹,移植到另外的田地上,或是向蕉苗場購買利用組織培養產生出來的香蕉苗。無論用哪一種方式栽種,母株和子株的基因幾乎是一樣的,保障香蕉的優良品種。
但,保留品種的反面,即缺乏基因多樣性。一旦現存品種對於某種疾病沒有抵抗力,即將面臨全面性的滅種危機……
黃葉病:香蕉的瘟疫
香蕉大滅絕,沒有香蕉吃——這可不是危言聳聽,而是真實發生過。一百多年前中南美洲主要栽種的香蕉品種是「大米七」,雖然外皮比較厚,但是香氣濃郁,果肉超甜,據說比現在的華蕉更美味。 1900 ~ 1960 年間,香蕉黃葉病肆虐,導致大米七幾近滅絕。
香蕉黃葉病又稱「巴拿馬病」,是一種叫做「尖孢鐮刀菌古巴專化型」的真菌所引起,這種真菌能以(厚壁)孢子的型態在土壤裡存活超過三十年,經由土壤、灌溉水或附著在農具或車輛上,四處傳播、感染栽種在田裡的香蕉。罹病的香蕉會由下方老葉葉緣先黃化,然後逐漸擴大,最後整株枯萎死亡。罹病的香蕉植株如果隨意丟棄,也會傳播病菌,難以防治。
所幸,後來可抗黃葉病的華蕉取代了大米七,成為目前全世界主要栽種的品種,人們才又有香蕉可以吃,臺灣的主力品種「北蕉」就是屬於華蕉。
然而,黃葉病的威脅並沒有遠離。1967 年台灣屏東縣佳冬地區的蕉園首次出現熱帶第四型黃葉病,罪魁禍首是尖孢鐮刀菌古巴專化型的第四型生理小種。華蕉雖然可以抵抗舊型的黃葉病,卻無法抵抗這種新型黃葉病。1990 年代,新型黃葉病繼續入侵東南亞,2013 年證實已入侵南亞、中東、非洲及澳洲的香蕉園!
香蕉研究所,培育抗黃葉病品種
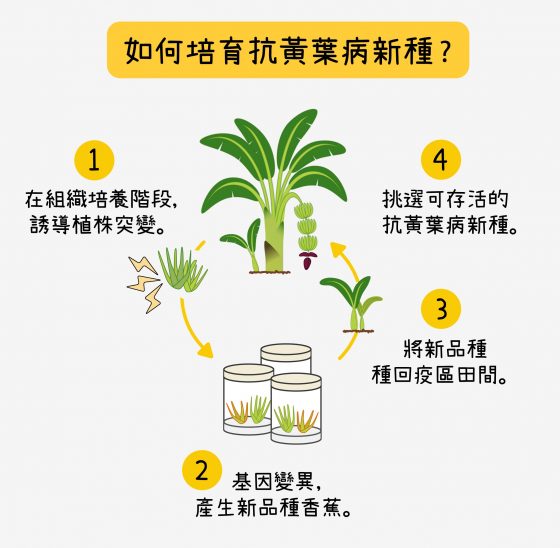
至此,全世界無不想辦法拯救香蕉的滅種危機,其中包括培育能抵抗新型黃葉病的香蕉新種,例如:台灣香蕉研究所培育的寶島蕉(又稱台蕉四號)、台蕉五號和七號,其中台蕉五號在台灣、寶島蕉在菲律賓皆已有商品化,除了抗病,也直接有經濟產值。一旦華蕉全面性的毀滅降臨時,這些可抗新型黃葉病的品種將會成為蕉農的救星。因為尖孢鐮刀菌很難用殺真菌劑殺死,又能潛藏在土壤中數十年,難以從田地中清除,改種抗病品種可說是解除黃葉病威脅最好的策略之一。
資料來源│蘇柏諺 (陳荷明實驗室)
圖說重製│林洵安
問題來了!未來如果將抗病品種推廣到其他國家,必須防範別的國家買了少量香蕉幼苗後,利用組織培養大量盜用。但過往作物多由植株或果實的外型做為分類,不容易分辨,爭議時也較難舉證。
為此,陳荷明研究團隊發揮基因序列資料分析的專長,找到了寶島蕉、台蕉五號和七號 DNA 上特殊的序列,一旦有人盜用,就能從這些獨特的 DNA 序列,也就是分子標誌,鑑定出該品種是否源自台灣香蕉研究所。
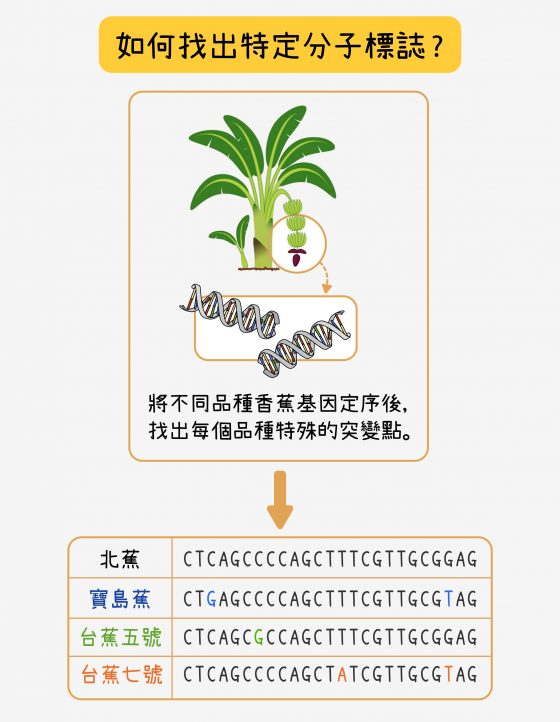
香蕉的身分證:具專一性的分子標誌
想要正確區隔出品種,分子標誌必須有專一性,以達到可信任的辨別度。陳荷明的研究團隊先把香蕉會表現的基因定序出來,利用電腦程式比對北蕉和抗病品種,找出哪些 DNA 序列不一樣,將有差異的序列一個個挑出來,進一步比對各個品種之間的差別,從中選出單一品種特有的分子標誌,作為鑑定品種的依據。
資料來源│蘇柏諺 (陳荷明實驗室)
圖說重製│林洵安
找到各個品種特有的分子標誌之後,陳荷明的團隊開發出兩種鑑定分子標誌的方法。
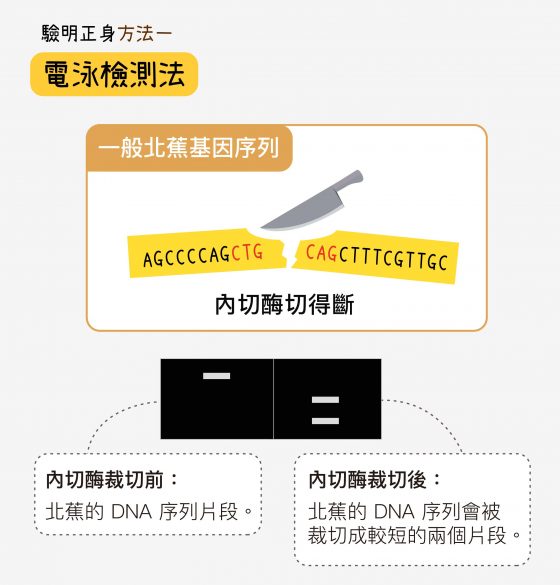
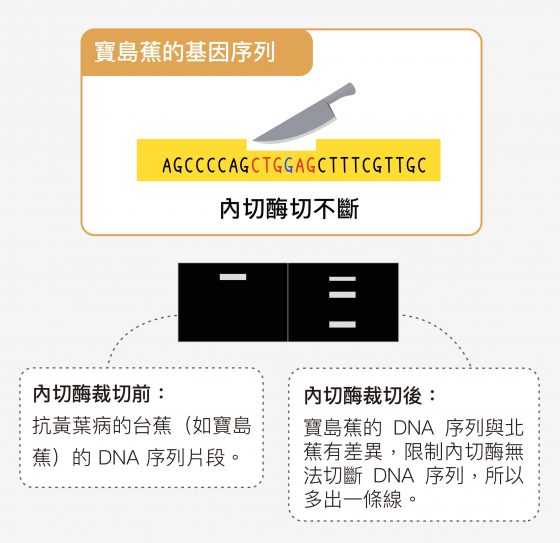
方法一:使用限制內切酶切割特定 DNA 片段。限制內切酶可與特定的 DNA 序列結合,將序列從中間切斷。它的專一性很高,如果序列有一點點不一樣,它就沒辦法結合、切斷序列。再用電泳法分離切割後的 DNA 片段,片段越小跑得越遠,如此一來,從電泳圖上條帶的數量和位置即可鑑定品種。
實際做法是:挑選一個 DNA 片段,如果受測基因序列屬於一般北蕉(不抗新型黃葉病),片段會被切斷,電泳會出現兩條線(代表被切斷後兩個較小片段)。如果這段基因序列屬於抗新型黃葉病的台蕉(如寶島蕉),片段無法被切斷,就會多出一條代表完整片段的線。這種方法費用相對便宜,而且不到三個小時就能知道結果。但不是所有找到的品種特有序列,皆能找到適當的限制內切酶來分辨。
資料來源│蘇柏諺 (陳荷明實驗室)
圖說重製│林洵安
資料來源│蘇柏諺 (陳荷明實驗室)
圖說重製│林洵安
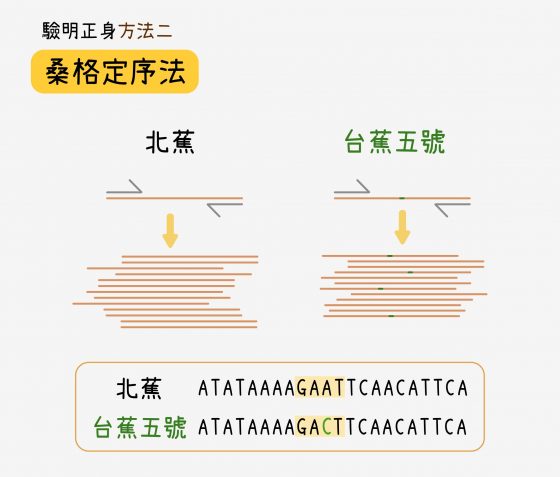
方法二:經由聚合酶連鎖反應大量複製分子標誌的 DNA 片段,然後以「桑格定序法」(Sanger sequencing),進行專一性片段的定序。簡言之,桑格定序能直接檢測出 DNA 片段上 ATCG 四種鹼基的排列順序(一般交由提供定序服務的廠商處理),檢查序列上特定位置是否有變異,就可以確認品種。
桑格定序法費用較高,定序時間比較久,但準確性高,目前陳荷明提供香蕉研究所的品種特有分子標誌,都能以桑格定序來檢測。
資料來源│蘇柏諺 (陳荷明實驗室)
圖說重製│林洵安
創新的檢驗方式,研發開端可謂篳路藍縷,除了要整合多種現有序列分析工具,嘗試不同參數設定,甚至得自行編寫程式!由於要比對的序列非常龐大,沒辦法用人工逐一比對,必須靠電腦運算。不是資訊背景出身的研究助理侯博瀚,自學程式設計,編寫程式比較北蕉與抗病品種間序列差異,實驗才能進行。之後,博士後江明豪和研究助理蘇柏諺針對找到的北蕉與台蕉差異序列,以多個在地及國際常見香蕉品種,進行廣泛地測試,終於驗證分子標誌的專一性。
那麼,陳荷明團隊的下一步呢?「既然找到抗病品種的特殊序列,是否能從其中找到抗黃葉病的基因呢?」事實上,確認抗病品種的分子標誌來區分香蕉品種,只是最基礎的工作。陳荷明和許多與黃葉病對抗的科學家正分頭努力,最終希望找到香蕉抗黃葉病的基因,掌握抗病的機制,幫助香蕉擊退黃葉病,讓蕉農有香蕉可種,民眾永遠有美味又方便的香蕉可享用。