- 作者 / 照護線上編輯部
- 本文轉載自 Care Online 照護線上《及早發現,把握黃金治療期,新生兒基因檢測醫師圖文解說》,歡迎喜歡這篇文章的朋友訂閱支持 Care Online 喔

在少子化的台灣,每個孩子都是寶,父母親殷切期盼孩子出世,更希望孩子能健康長大。隨著檢測技術的進步,家長們也有越來越多的選擇。
最首要的是國家提供的篩檢項目,包含代謝篩檢與聽力篩檢。目前的新生兒先天性代謝異常疾病篩檢有 21 個項目,這些代謝疾病若未能及時治療,可能造成損害,導致永久性後遺症。禾馨醫療小兒科陳菁兒醫師指出,國民健康署提供的新生兒篩檢主要是篩檢「採檢當下」血液中的代謝產物、酵素,可以找出急性或嚴重程度較高的疾病,以便即早接受治療、減輕症狀。至於聽力篩檢,可以找出當下已經發生嚴重聽力損傷的寶寶。
由政府補助的各項篩檢皆相當重要,但還是有其侷限性,陳菁兒醫師分析,所以有些未列入篩檢的疾病,或者是比較晚發病的寶寶,往往得等到出現症狀、生長遲緩時,才就醫檢查,可能錯過黃金治療期,而造成父母的自責與遺憾。
受惠於醫學的進展,目前已可運用基因檢技術讓新生兒篩檢更加完善,「新生兒基因檢測」只需寶寶採取幾滴血液或是刮取口腔黏膜細胞便能進行檢驗,能夠提早驗出晚發型的寶寶,且涵蓋更多疾病種類。
新生兒聽力篩檢正常,長大後卻出現聽力損傷!?
新生兒基因檢測的起源與聽力篩檢有關,陳菁兒醫師解釋,以往大家認為只要通過新生兒聽力篩檢的寶寶,聽力應該就沒有問題,但是後來卻發現,部分在 4-5 歲有語言發展遲緩的小朋友,檢查起來其實是聽力損傷造成的學習遲緩,代表仍有部份聽力損傷無法透過新生兒聽力篩檢出來。
台大醫院耳鼻喉科醫師與基因醫學部團隊針對這個問題深入研究,這才發現台灣人具有一些特別常見的聽損基因及點位,於是發展出聽損基因檢測在新生兒的應用。
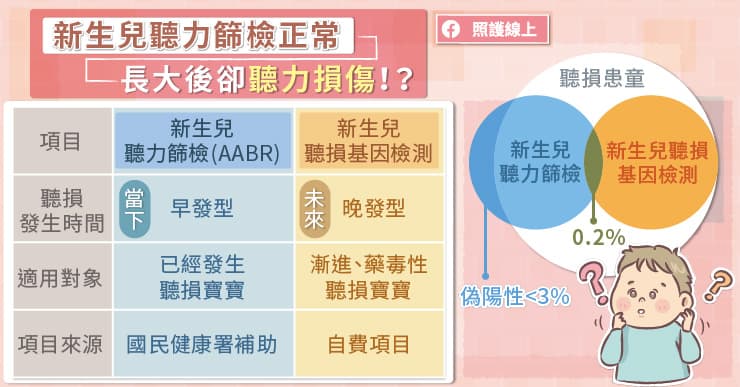
陳菁兒醫師說明,上圖中藍色圈圈代表由新生兒聽力篩檢所發現當下已經有嚴重聽損的寶寶,橘色圈圈是利用基因檢測所找出來較輕微、以及晚發型的聽損寶寶。研究發現這兩種檢查重疊的部分很小,也就是說新生兒基因檢測可以找到傳統聽力篩檢無法篩檢到的族群,且兩者同等重要,無法互相取代。
這樣的研究開啟了新生兒基因篩檢的應用,在台灣也已經頗為普及,準父母在產檢過程中都可以由醫療院所獲得相關資訊。
新生兒基因檢測涵蓋更廣泛
不同檢測技術,可以找出的不同疾病特性的族群,陳菁兒醫師說明,「新生兒代謝篩檢」所檢驗的採檢當下寶寶血液中的代謝物質,可以偵測出急性且嚴重的代謝疾病,但輕型或晚發型代謝疾病就不一定驗得出來;「新生兒基因檢測」則可以提早找出高風險寶寶,後續便能加強照護和追蹤。
除了聽力損傷及代謝疾病,目前新生兒基因檢測的應用範圍很廣,還可以進階到找出特定中樞神經、血液、肌肉、心臟、視力等多種疾病,陳菁兒醫師說,透過基因檢測甚至可以了解一些藥物過敏的問題。

「基因檢測可以實現預防醫學與精準醫療的概念。」陳菁兒醫師說,「以藥物過敏為例,輕微的可能只是皮膚搔癢紅腫,但嚴重可能導致死亡。過去大家只能在用藥之後,才從臨床反應知道是否對藥物過敏,現在則可利用藥物基因檢測找出可能引起過敏反應的藥物,就醫時就能提醒醫師避免使用相關藥物,或調整藥物劑量,以降低藥物不良反應發生的機會。」
藥物基因體學包括藥物代謝與藥物不良反應的資訊,陳菁兒醫師分析,因為每個人對藥物代謝的速率不同,所以在相同劑量下可能產生不同的反應,譬如小朋友常用的退燒藥,可能因為代謝速率不同而導致不良反應,嚴重可能造成腸胃道出血。成人常用的降尿酸藥物,在某些人身上可能出現史帝芬強生症候群 (Steven-Johnson Syndrome,SJS) 或毒性上皮溶解症 (Toxic Epidermal Necrolysis,TEN),皮膚會嚴重潰爛,甚至導致死亡。
「還有知名電視劇「麻醉風暴」中上演的惡性高熱,主要是因為帶有特殊基因的人群,在使用某些麻醉藥物後容易演變成惡性高熱,而危及性命。」陳菁兒醫師說,「以往這些比較特殊的案例,常找不到原因,但是現在透過藥物基因檢測,我們可以提早找出容易對這些高敏感藥物產生不良反應的族群,有助於提升用藥安全。」
因為人體的遺傳基因不會改變,一輩子只需要驗一次即可,這些攸關用藥安全的資訊,長大以後也都非常實用。
基因檢測讓家族成員連帶受益
針對有家族史的寶寶,因為帶有相同疾病基因變異的機率比較高,需要以基因檢測確認是否為高風險寶寶,提早做預防照護。
至於沒有家族史的寶寶,則可以透過基因檢測找出相關疾病,並能藉此找出隱藏的家族史而讓整個家族受益。
也就是說,新生兒基因檢測的好處其實不只侷限在受檢的寶寶,陳菁兒醫師分析,包含已出生、未出生的兄弟姊妹、其他家族成員,甚至上一代或下一代都可能是受益的對象。
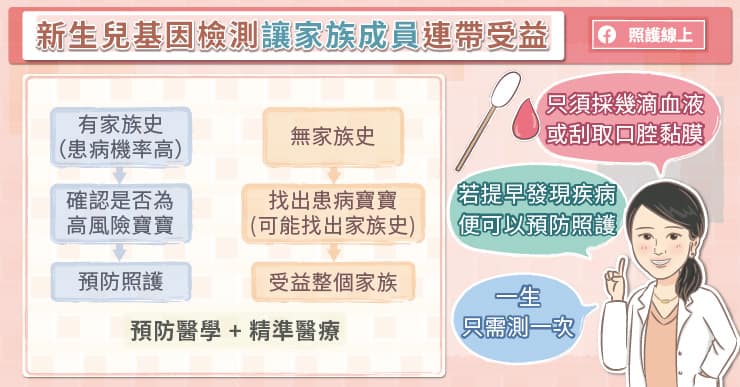
無論是否有已知家族史,如果新生兒驗出疾病基因變異,都應該接受完整遺傳諮詢,必要時再進行相對應基因檢測。「有的家族因為寶寶做了基因檢測,才發現其他家族成員其實也有相同的疾病,因而得以提早接受治療。」陳菁兒醫師說,「另外,了解遺傳疾病的狀況,也可以讓爸爸媽媽在準備生育下一胎時,做好計畫。」
貼心小叮嚀
陳菁兒醫師提醒,基因檢測是一種風險評估的概念,一生只需驗一次,就可以得到終身受用的基因疾病資訊。提早檢測就越能夠提早預防,避免錯失疾病的黃金治療期,甚至其他家族成員也可能連帶受益喔!
- 本文轉載自 Care Online 照護線上《及早發現,把握黃金治療期,新生兒基因檢測醫師圖文解說》,歡迎喜歡這篇文章的朋友訂閱支持 Care Online 喔
- 加入照護線上 LINE 官方帳號,健康資訊不漏接





















 標靶藥物未發揮效果或抗藥後 可利用次世代基因定序(NGS)找線索
標靶藥物未發揮效果或抗藥後 可利用次世代基因定序(NGS)找線索 她的故事 談自我照顧
她的故事 談自我照顧